Fonctionnement et performances d'across dans dplyr
A la sortie de l'été dernier, j'ai réalisé une note sur mon blog personnel sur un élément important d'une mise à jour majeure de dplyr : across(), un nouveau verbe pour réaliser des opérations sur plusieurs colonnes. Dans cette note, on utilisait la version 1.0.2 de dplyr et on comparait across aux verbes équivalents que cela devait remplacer (fonctions indexées par _at, _if et _all) . On constatait une moins bonne performance d' across en termes de temps d'exécution. Cet élément était bien connu des développeurs de RStudio et a été constamment pris en compte dans les différentes mises à jour. On reprend ici cette note en la mettant à jour avec la version 1.0.6 de dplyr disponible à ce jour pour voir où se place désormais across en termes de temps d'exécution.
Si vous voulez balayer plus largement les différents éléments de la mise à jour de dplyr, vous pouvez vous rendre sur le site du tidyverse (en anglais) ou sur cet article du blog de ThinkR (en français) qui en présentent les changements majeurs.
across(), ça marche comment?
Syntaxe de base
Le verbe across() vise à remplacer toutes les fonctions suffixées par _if, _at et _all. Il regroupe ces méthodes dans une seule et permet ainsi de les associer, ce qui n'était pas possible avant. Il s'utilise dans mutate et summarise. La syntaxe associée à ce verbe est la suivante :
across(.cols, .fns)
Dans laquelle :
- Les colonnes .cols peuvent être sélectionnées en utilisant la même syntaxe que pour la méthode vars() (nom des variables, starts_with, end_with, contains,...), mais aussi avec des conditions rentrées dans where() qui sélectionneront de la même manière que le faisaient les fonctions suffixées par _if.
- La fonction .fns est définie comme auparavant (le nom de la fonction ou sa définition "à la volée" avec ~ my_fun(.)).
On présente quelques exemples en utilisant la table penguins promue par Allison Horst pour remplacer l'usage de la table iris. Vous pouvez l'obtenir depuis le package palmerpenguins sur le CRAN.
Sélection avec des conditions
À partir de cette table, l'instruction visant à sortir la moyenne de toutes les variables numériques s'écrivait auparavant :
penguins %>% summarise_if(is.numeric, mean, na.rm = TRUE)
## # A tibble: 1 x 5
## bill_length_mm bill_depth_mm flipper_length_mm body_mass_g year
## <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 43.9 17.2 201. 4202. 2008.
Elle se réécrit avec across() en utilisant where() :
penguins %>% summarise(across(where(is.numeric), mean, na.rm = TRUE))
## # A tibble: 1 x 5
## bill_length_mm bill_depth_mm flipper_length_mm body_mass_g year
## <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 43.9 17.2 201. 4202. 2008.
Sélection à partir du nom
Si l'on souhaite sélectionner à partir du nom des variables, la nouvelle syntaxe est la suivante :
# Ancienne version
penguins %>% summarise_at(vars(matches("bill*|flipper*")), mean, na.rm = TRUE)
## # A tibble: 1 x 3
## bill_length_mm bill_depth_mm flipper_length_mm
## <dbl> <dbl> <dbl>
## 1 43.9 17.2 201.
# Avec across()
penguins %>% summarise(across(matches("bill*|flipper*"), mean, na.rm = TRUE))
## # A tibble: 1 x 3
## bill_length_mm bill_depth_mm flipper_length_mm
## <dbl> <dbl> <dbl>
## 1 43.9 17.2 201.
Autres propriétés
On note également qu'on peut combiner dorénavant les sélections sur les types des colonnes et sur leur nom dans une seule instruction across(), ce qui n'était pas possible avant. Pour enlever les années des moyennes numériques, on peut par exemple écrire :
penguins %>% summarise(across(where(is.numeric) & -contains("year"), mean, na.rm = TRUE))
## # A tibble: 1 x 4
## bill_length_mm bill_depth_mm flipper_length_mm body_mass_g
## <dbl> <dbl> <dbl> <dbl>
## 1 43.9 17.2 201. 4202.
Enfin, le paramètre .names de across() est également très pratique et permet notamment dans une instruction mutate() de créer de nouvelles colonnes nommées à partir des anciennes auxquelles on peut se référer avec .col. Par exemple, si je veux créer deux nouvelles colonnes passant les informations sur le bec en pouces mais en conservant les anciennes colonnes, je peux écrire :
penguins %>%
mutate(across(starts_with("bill"), ~ . * 0.04, .names = "pouces_{.col}")) %>%
select(contains("bill")) %>% head(5)
## # A tibble: 5 x 4
## bill_length_mm bill_depth_mm pouces_bill_length_mm pouces_bill_depth_mm
## <dbl> <dbl> <dbl> <dbl>
## 1 39.1 18.7 1.56 0.748
## 2 39.5 17.4 1.58 0.696
## 3 40.3 18 1.61 0.72
## 4 NA NA NA NA
## 5 36.7 19.3 1.47 0.772
across(), ça tourne comment?
À la sortie de la mise à jour de dplyr, il avait été signalé que la méthode across() impliquerait peut-être de légères pertes en termes de vitesse d'exécution par rapport aux anciennes méthodes _at, _if et _all. On a mis en évidence ce problème avec la version 1.0.2 de dplyr dans la première version de cet article. Sur ce même modèle, on va comparer les instructions _if et _at d'un summarise groupé avec leurs équivalents dans across() pour différentes tailles d'échantillons et de groupes.
Instructions sur lesquelles on compare les méthodes
On crée un tibble comportant 4 variables numériques et une variable facteur, et on va comparer la vitesse d'exécution des moyennes de ces variables numériques groupées par modalité de la variable facteur en faisant varier le nombre de lignes du tibble et le nombre de groupes (de modalités distinctes de la variable facteur). Le tibble est créé par exemple ainsi, pour 100 lignes et deux groupes :
nbrow <- 100
nbgpe <- 2
as_tibble(data.frame(x1 = rnorm(nbrow), x2 = rnorm(nbrow),
x3 = runif(nbrow), x4 = runif(nbrow),
y = as.factor(sample(floor(nbgpe), replace = TRUE))
)) %>%
arrange(x1) %>%
mutate(across(where(is.numeric), ~ round(., 2)))
## # A tibble: 100 x 5
## x1 x2 x3 x4 y
## <dbl> <dbl> <dbl> <dbl> <fct>
## 1 -3.36 0.9 0.32 0.36 2
## 2 -3.04 0.92 0.61 0.46 2
## 3 -2.4 1.37 0.32 0.98 2
## 4 -2.08 1.64 0.87 0.72 2
## 5 -2.05 1.22 0.62 0.79 2
## 6 -1.78 -1.28 0.58 0.31 2
## 7 -1.56 1.19 0.96 0.58 2
## 8 -1.55 -1.18 0.4 0.85 2
## 9 -1.47 1.77 0.71 0.21 2
## 10 -1.29 -0.6 0.68 0.4 2
## # … with 90 more rows
Sur chaque tibble (chaque combinaison du nombre de lignes et de groupes), les différentes instructions testées sont les suivantes :
# summarise_if
data %>% group_by(y) %>% summarise_if(is.numeric, mean)
# across + where()
data %>% group_by(y) %>% summarise(across(where(is.numeric), mean))
# summarise_at
data %>% group_by(y) %>% summarise_at(vars(starts_with("x")), mean)
# across + starts_with()
data %>% group_by(y) %>% summarise(across(starts_with("x"), mean))
Toutes les instructions font la même chose : une moyenne groupée par y des 4 variables numériques. L'idée est de vérifier que l'option across n'est pas plus lente que les options summarise_if et summarise_at.
Résultats de la version 1.0.6
Les résultats du microbenchmark() pour les différentes combinaisons de nombres de groupes et de lignes sont présentés dans un graphique qui représente la distribution du temps d’exécution des 10 occurences testées pour chaque méthode :
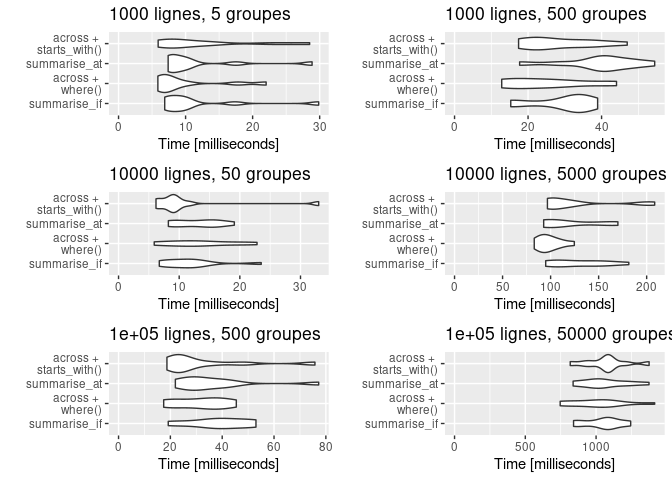
Sur nos exemples, il semblerait qu'across ait complètement rattrapé son retard sur ses équivalents _at et _if. Il semble même légèrement plus performant dans certains cas de figure.
Résultats de la version 1.0.0
Pour illustrer le chemin parcouru, on peut refaire tourner cette même comparaison avec la version 1.0.0 de dplyr :

On constate bien que dans sa première version, across connaissait de bien moins bonnes performances, en particulier sur les dataframes avec beaucoup de lignes et/ou beaucoup de groupes. Les mises à jour successives ont donc bien permis de combler ces problèmes de performance et c'est une excellente nouvelle car au niveau de la syntaxe, nous, on adore!
C'est tout pour aujourd'hui ! Comme d'habitude vous pouvez retrouver le fichier Rmarkdown ayant servi à générer cette note sur le github de Statoscop.